Investigador da U.Porto ajuda a desvendar árvore filogenética das aves
Descoberta publicada na revista "Nature" lança
as bases para a construção da árvore genealógica de aves mais abrangente
de sempre.

Depois de décadas refém de resultados contraditórios, a árvore filogenética das aves acaba finalmente de ser desvendada, com a colaboração
do investigador do Centro Interdisciplinar de Investigação Marinha e
Ambiental (CIIMAR) e professor da Faculdade de Ciências da Universidade
do Porto (FCUP), Agostinho Antunes.
Muitos estudos anteriores tinham tentado construir a árvore
filogenética das aves, usando pequenos conjuntos de dados de diferentes
regiões genómicas. Contudo, estes trabalhos produziam frequentemente
resultados contraditórios.
O estudo intitulado “Complexity of avian evolution revealed by family-level genomes”, publicado na revista prestigiada Nature e que envolveu o investigador do CIIMAR e professor associado
FCUP, permitiu usar, pela primeira vez, dados completos a uma escala
genómica para construir a árvore para espécies de aves. Uma reconstrução
intrincada que ilustra 93 milhões de anos de relações evolutivas entre
363 espécies de aves, representando 92% de todas as famílias destes
animais.
Segundo Agostinho Antunes, “esta nova árvore genealógica será uma
base sólida para mapear a história evolutiva de todas as espécies de
aves, com implicações importantes para a investigação ornitológica,
estudos de biodiversidade e conservação.”
Este conjunto completo de dados genómicos foi produzido pelo consórcio B10K (Bird 10 000 Genomes Project) após a sequenciação de 48 genomas de aves publicados em 2014 na revista Science,
que também contou com a colaboração de investigador do CIIMAR e
professor da FCUP. O trabalho publicado agora efetua um grande avanço
aos trabalhos anteriores.
Mais dados e mais completos
Ao empregar dados completos do genoma de 363 espécies de aves, o
estudo agora publicado envolveu o maior conjunto de dados já utilizado
para análises filogenéticas de aves. Para isso, a equipa do B10K construiu um novo pipeline
para extrair mais de 150 mil regiões espalhadas pelo genoma. A
caracterização das relações filogenéticas em todo o genoma permitiu
identificar padrões associados ao contexto genómico e às características
da sequência.
Agostinho Antunes clarifica de que forma isso distingue este estudo
das tentativas anteriores: “As várias partes do genoma, por exemplo,
cromossomas individuais ou genes codificadores de proteínas, sustentam,
muitas vezes, árvores muito diferentes. Isso possivelmente explica o
motivo pelo qual os estudos anteriores, que analisaram apenas certas
porções genómicas, estavam em conflito.”
A equipa de trabalho descobriu que, para a maioria dos agrupamentos, é
possível chegar a um consenso sobre os seus relacionamentos, quando uma
quantidade suficiente de dados é usada. Mas as posições filogenéticas
de alguns grupos de aves, como é o caso das corujas ou dos falcões,
permanecem intrigantes, mesmo com dados em escala completa do genoma. É
nestes casos, em que reunir grandes quantidades de dados de alta
qualidade é fundamental para se produzir uma árvore filogenética
robusta.
Esta nova árvore genealógica resolve alguns destes debates de longa
data sobre as relações entre as espécies de aves e estabelece uma base
sólida para o estudo da evolução das aves e do desenvolvimento de
características, desvendando novos caminhos naquela que ainda é a longa
jornada para compreender completamente os mistérios da evolução das
aves.

Professor Associado do Departamento de Biologia da FCUP, Agostinho Antunes lidera o grupo de «Genómica Evolutiva e Bioinformática» do CIIMAR
Uma escala temporal precisa
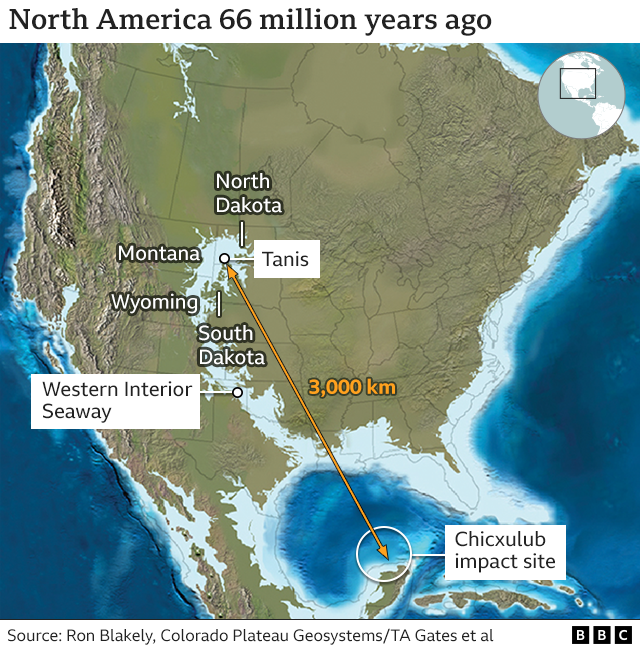
Apesar dos esforços anteriores para compreender os impactos do evento
Cretácico-Paleogénico, utilizando dados morfológicos e moleculares, as
relações entre as linhagens neoaviárias permaneceram controversas. Os
métodos aplicados neste trabalho basearam-se na comparação de genomas de
espécies vivas que, no entanto, permitem obter informação de eventos
que aconteceram há muito tempo atrás.
Através de métodos computacionais de ponta e recursos de
supercomputação de última geração, o estudo permitiu propor uma escala
temporal mais precisa para a diversificação das aves modernas, sugerindo
que se deu uma rápida radiação durante ou perto da extinção em massa na
fronteira Cretácico-Paleogénico (o evento associado à extinção dos
dinossauros) e após o Paleogénico-Neogénico.
“Aproximadamente 95% de todas as espécies de aves atuais (Neoaves),
emergiu desta radiação!” reforça o investigador do CIIMAR e professor na
FCUP. A nova árvore desafia a organização dos Neoaves ao classificar
este grande grupo em quatro clados principais: Mirandornithes,
Columbaves, Elementaves e Telluraves.
Os investigadores descobriram que estas radiações coincidiam com
mudanças genéticas e morfológicas notáveis dentro das aves: maiores
taxas de mutação, tamanhos corporais menores, cérebros maiores e
tamanhos populacionais efetivos maiores.
“As aves são a única linhagem de dinossauros que sobreviveu até hoje.
Há cerca de 66 milhões de anos, na fronteira do Cretácico-Paleogénico, um
evento de extinção em massa destruiu todos os dinossauros não-aviários,
proporcionando uma oportunidade para as aves se diversificarem
rapidamente e ocuparem uma ampla gama de nichos ecológicos. As neoaves,
um grupo diversificado que compreende aproximadamente 95% de todas as
espécies de aves atuais, emergiu desta radiação. Dos imponentes condores
dos Andes aos diminutos colibris que voam pelas florestas tropicais, as
neoaves abrangem uma impressionante diversidade de formas e funções.”,
conta Agostinho Antunes.
As primeiras aves
Usando métodos computacionais avançados, os investigadores também
conseguiram esclarecer algo invulgar que tinha sido descoberto em 2014,
mas que nunca tinha sido devidamente compreendido: uma secção específica
de um cromossoma no genoma das aves permaneceu inalterada durante
milhões de anos, anulando os padrões esperados de recombinação genética.
“Esta anomalia foi baseada na análise dos genomas de apenas 48
espécies de aves e levou inicialmente os investigadores a agrupar
incorretamente os flamingos e as pombas como grupos evolutivos próximos,
pois pareciam intimamente relacionados com base nesta secção inalterada
do DNA. Ao repetir a análise usando os genomas de 363 espécies do atual
estudo, surgiu uma árvore genealógica mais precisa que afastou os
pombos dos flamingos. Essa deteção permitiu identificar atualmente um
evento preciso que terá acontecido há mais de 60 milhões de anos, o que é
extraordinário” explica o investigador do CIIMAR.
Este avanço é detalhado num artigo complementar publicado no Proceedings of the National Academy of Sciences (PNAS)
. Neste trabalho, os investigadores examinaram de perto um dos ramos da
nova árvore genealógica e descobriram que os flamingos e as pombas
estão mais distantemente relacionados do que as análises anteriores do
genoma tinham mostrado.
Os resultados destes trabalhos lançam assim uma nova luz sobre os
mecanismos adaptativos que impulsionaram a diversificação aviária no
rescaldo do evento de extinção em massa Cretácico-Paleogénico.
Um voo de longa duração
Avançando com a investigação, a equipa promete continuar o esforço
para desvendar por completo a evolução das aves. O trabalho futuro
pretende conciliar a sequenciação dos genomas de outras espécies de aves
para expandir a árvore genealógica a milhares de géneros de aves e
optimizar algoritmos e recursos computacionais para acomodar conjuntos
de dados ainda maiores, a fim de garantir que as análises em estudos
futuros sejam conduzidas com alta velocidade e precisão.
No entanto, já é possível dizer que estes resultados alteram
imensuravelmente as visões tradicionais sobre a história evolutiva das
aves. Esta nova árvore genealógica será uma base sólida para mapear a história evolutiva de todas as espécies de aves, com implicações importantes para a investigação ornitológica, estudos de biodiversidade e conservação.
Como destaca Agostinho Antunes, os métodos computacionais
desenvolvidos irão certamente ajudar a clarificar a árvore genealógica e
a história evolutiva de outros seres vivos: “O impacto deste trabalho
vai muito além do estudo da história evolutiva das aves. Os métodos
computacionais pioneiros desenvolvidos irão certamente tornar-se
ferramentas padrão para reconstruir árvores evolutivas de uma grande
variedade de outros animais.”
Sobre o projeto B10K
O Projeto B10K (Bird 10 000 Genomes) é uma iniciativa que visa mapear
os genomas de todas as aproximadamente 10.500 espécies de aves
atualmente existentes na Terra.
Este ambicioso projeto procura construir uma árvore da vida das aves
abrangente a partir do conhecimento completo dos seus genomas,
descodificando as ligações entre a variação genética e as diferenças de
características fenotípicas, desvendando a evolução molecular, a
biogeografia e as inter-relações da biodiversidade, avaliando o impacto
das mudanças ambientais e humanas, atividades sobre evolução das
espécies e biodiversidade, e revelando a história populacional de todo o
grupo de aves.
Os trabalhos agora publicados do B10K foram lideradas por Josefin Stiller (University of Copenhagen, Denmark), Siavash Mirarab (University of California San Diego, USA) e Guojie Zhang (Zhejiang University, China), e incluíram vários outros investigadores internacionais entre os quais Agostinho Antunes.
in Notícias Universidade do Porto








.jpg)
.jpg)
.jpg)